データのダウンロード
- http://usegalaxy.eu/ にログインする。US版
- 新しいヒストリーを作成し、「AutoDock」という名前を付ける。
- ARとDHTの複合体の立体構造をダウンロードする。
- ツールパネルからGet PDBを選ぶ。
- “PDB accession code” に
3L3Xを入力する。
- Run Toolボタンを押して実行する。
- データの名前を
AR structureに変更する。
- 候補化合物のデータをアップロードする。
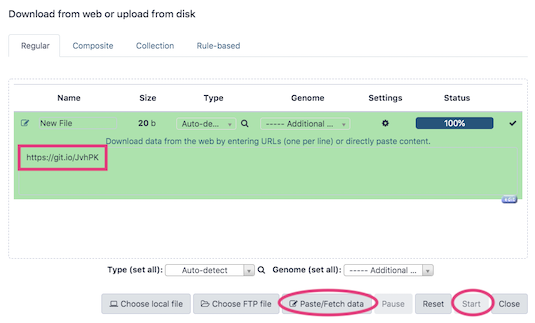
- 「Download from web or upload from disk」ダイアログの下部にあるPaste/Fetch dataを押す。
- 出現したボックスにデータのURL
https://git.io/JvhPK を入力し、Startを押す。
- データの名前を
Compound libraryに変更する。
前処理(タンパク質)
- ARの立体構造を取り出す。
- ツールパネルからSearch in textfiles (grep)を選ぶ。
- “Select lines from”: 先ほどダウンロードした
AR structure
- “that”:
Don't match
- “Regular Expression”:
HETATM
- Executeボタンを押して実行する。
- データの名前を
Protein (PDB)に変更する。
- (オプショナル)DHTの立体構造を取り出す。
- ツールパネルからSearch in textfiles (grep)を選ぶ。
- “Select lines from”: 先ほどダウンロードした
AR structure
- “that”:
Match
- “Regular Expression”:
DHT
- Run Toolボタンを押して実行する。
- データの名前を
Ligand (PDB)に変更する。
- Autodock Vinaに入力できるファイルへ変換する。
- ツールパネルからPrepare receptorを選ぶ。
- “Select a PDB file”:
Protein (PDB)
- Run Toolボタンを押して実行する。
- データの名前を
Protein (PDBQT)に変更する。
前処理(候補化合物)
- 候補化合物の前処理を行う。
- ツールパネルからCompound conversionを選ぶ。
- “Molecular input file”:
Compound library
- “Output format”:
MDL MOL format (sdf, mol)
- “Generate 3D coordinates”:
Yes
- Run Toolボタンを押して実行する。
- データの名前を
Prepared ligandsに変更する。
ドッキング
- AutoDock Vinaによるドッキングを実行する。
- ツールパネルからVina Dockingを選ぶ。
- “Receptor”:
Protein (PDBQT)
- “Ligands”:
Prepared ligands
- “Specify pH value for ligand protonation”:
7.4
- “Specify parameters”:
Specify values directly
- “x coordinate”:
6, “y coordinate”: 30, “z coordinate”: 12
- “x size”:
56, “y size”: 56, “z size”: 72
- “Specify random seed for simulation reproducibility?”:
Yes
- “Random seed”: 1
- Run Toolボタンを押して実行する。
- 得られたドッキングのAffinityを取得する。
- ツールパネルからExtract values from an SD-fileを選ぶ。
- “Input SD-file”: ドッキングの結果(Collectionアイコンを選択すること)
- “Include the property name as header”:
Yes
- “Include molecule name as column in output”:
Yes
- Run Toolボタンを押して実行する。
- この結果を見ると、各候補化合物に対していくつかのドッキングが得られていることがわかる。SCORE列にAffinityが記されており、Affinity ≦ -8.0ならば結合すると見なす。ただし、実験対象とするタンパク質・化合物によっては必ずしも適切ではないが、本課題ではこの閾値を用いる。
可視化
- ドッキング結果をPDB形式に変換する。
- ツールパネルから
Compound conversionを選ぶ。
- “Molecular input file”: ドッキングの結果(Collectionアイコンを選択すること)
- “Output format”:
Protein Data Bank format (pdb)
- “Split multi-molecule files into a collection”:
Yes
- Run Toolボタンを押して実行する。
- 結果をダウンロードする。
- NGL Viewer (http://nglviewer.org/ngl/) で可視化する。
- ARの立体構造 (
Protein (PDB)) をダウンロードする。
- 見たいドッキング結果のPDBを上のコレクションの中からダウンロードする。
- NGL Viewerに上の二つのPDBをドロップする。

